La Universidad de Granada colabora en un estudio internacional, publicado en la revista Nature Genetics, que ha supuesto la secuenciación masiva de 81 especies de parásitos helmintos y la identificación de 6.000 familias de genes asociados a los mecanismos patogénicos involucrados en las enfermedades parasitarias. Más de 2.000 millones de personas sufren en el mundo enfermedades parasitarias, la mayoría en países pobres, y actualmente no existen vacunas para combatirlas.
Científicos de la Universidad de Granada (UGR) han participado en el mayor estudio genómico realizado hasta la fecha en parásitos helmintos, gusanos intestinales que causan enfermedades crónicas debilitantes en humanos y animales, diezman la producción de la ganadería y son un impedimento importante para el desarrollo socioeconómico de amplias zonas del planeta.
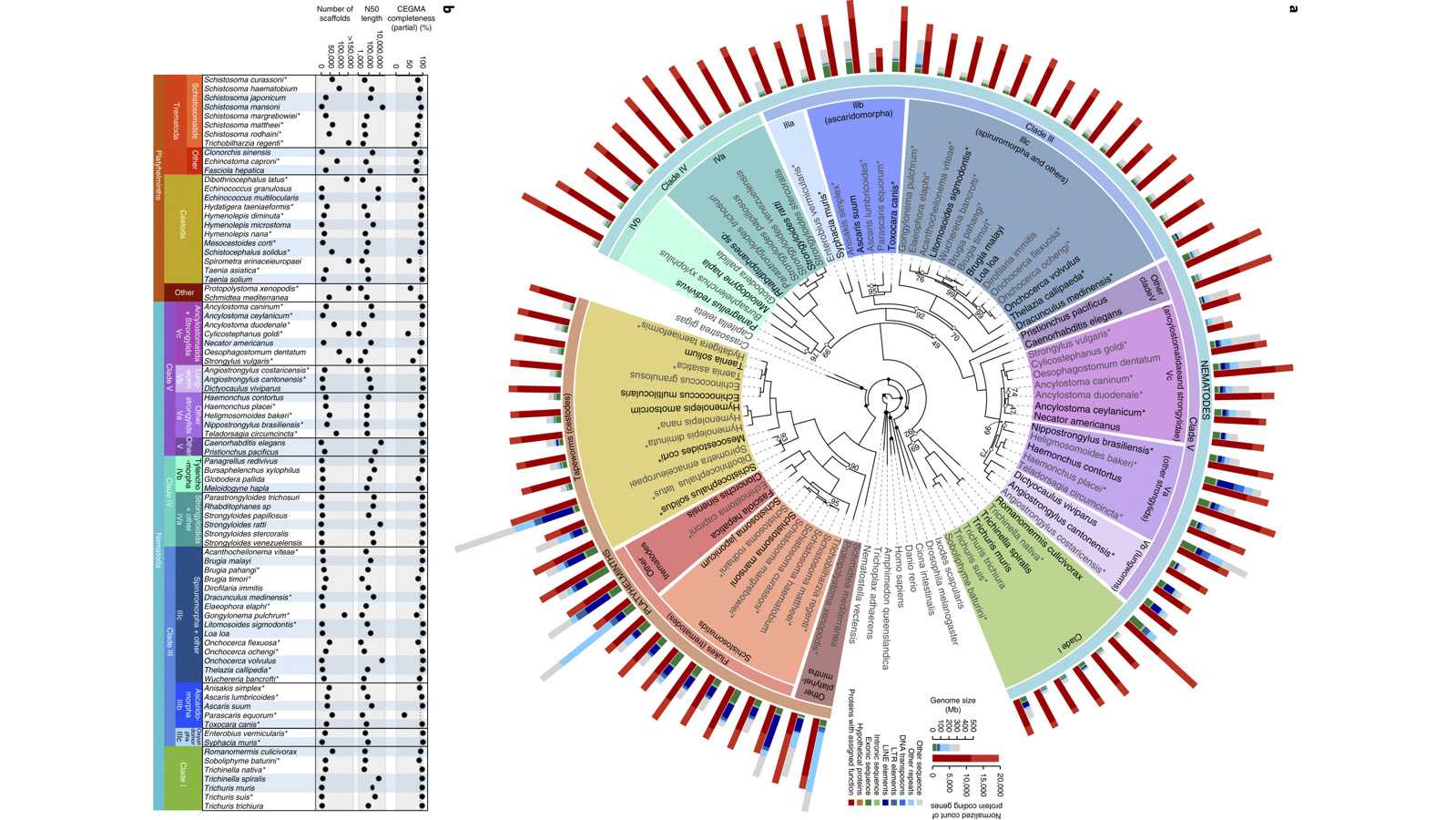
Este trabajo, realizado por un equipo internacional y coordinado por el Welcome Sanger Institute de la Universidad de Cambridge, ha supuesto la secuenciación masiva de 81 especies de helmintos y la identificación de 6.000 familias de genes asociados a los mecanismos patogénicos involucrados en las enfermedades parasitarias. Los resultados se han publicado en la prestigiosa revista Nature Genetics.
Las enfermedades producidas por estos helmintos o gusanos intestinales afectan a aproximadamente a más de 2.000 millones de personas en el mundo, y entre ellas se encuentran la Schistosomiasis, Teniasis, Hidatidosis o la Filariariasis.
Muchas de ellas se denominan enfermedades desatendidas u olvidadas, y afectan principalmente a las poblaciones más necesitadas. También son enfermedades prevalentes en animales, y producen enormes pérdidas económicas en el sector primario. En el momento actual no se dispone de vacunas para combatirlas y existen pocos fármacos disponibles para tratarlas, incluso con elevado riesgo de generar resistencias.
Rutas metabólicas
Desde el año 2011 los científicos participantes en este trabajo han llevado a cabo la secuenciación masiva de 81 especies de helmintos, cuyos genomas han sido anotados y comparados, prediciendo 1,6 millones de proteínas e identificando cerca de 6.000 familias de genes asociados con los mecanismos patogénicos involucrados en estas enfermedades.
También, y en base a la comparación de los 81 genomas, se ha construido un mapa con las rutas metabólicas que siguen estos parásitos determinando nuevas dianas de acción terapéutica para combatirlos. En este sentido, en la última parte del trabajo, se ha realizado un cribado in silico de más de 5.000 compuestos que ha posibilitado la identificación de nuevos fármacos activos frente a las patologías que provocan.

Los investigadores de la UGR que han participado en este trabajo internacional. De izquierda a derecha, Teresa Cruz, Antonio Osuna y Mercedes Gómez.
Durante los últimos ocho años, un total de 54 instituciones de 16 países -seis europeos, cuatro americanos, cuatro asiáticos, uno africano y otro en Oceanía- han participado en la investigación. Entre ellos se encuentran los investigadores de la UGR Antonio Osuna, Mercedes Gómez y Teresa Cruz, miembros del grupo de investigación de la Universidad de Granada “CTS 183 Bioquímica y Parasitología Molecular”, del departamento de Parasitología (Instituto de Biotecnología IBTUGR).
El catedrático de la UGR Antonio Osuna destaca que en este análisis “hemos encontrado una serie de nuevas familias génicas que participan en la evolución hacia la vida parásita. De igual manera, se han identificado y comparado las familias de genes involucradas en la evasión de las respuestas inmunitarias del huésped, los genes que codifican proteínas que permiten la migración del parásito a través de los tejidos del hospedador o permiten que el parásito se alimente”.
También se han identificado aquellos genes que pueden ser usados para el diseño y desarrollo de nuevos fármacos que actúen de forma específica frente a estos organismos parásitos.
“Los resultados de este estudio proporcionaran las herramientas moleculares para el estudio en la comunidad científica entienda y combata los gusanos parásitos tanto para el hombre como de los animales de interés económico”, destaca Osuna.
Referencias:
Comparative genomics of the major parasitic worms
International Helminth Genomes Consortium
Nature Genetics (2018) Nov 5. doi: 10.1038/s41588-018-0262-1.
Suscríbete a nuestra newsletter
y recibe el mejor contenido de i+Descubre directo a tu email