La nueva biblioteca, denominada inCREDBle, es una base de datos online que recoge el perfil genómico de 461 cepas de bacterias resistentes a antibióticos procedentes de 41 hospitales de 13 comunidades españolas. Este recurso ha sido posible gracias a una nueva metodología que permite obtener secuencias completas de manera más rápida y a gran escala.
Las bacterias resistentes a los antibióticos, causantes de infecciones respiratorias y urinarias, se han convertido en una preocupación para la salud pública en todo el mundo debido a su capacidad para desarrollar resistencia a los antibióticos más efectivos hasta la fecha, los carbapenémicos. Según la Organización Mundial de la Salud (OMS), como resultado de la resistencia a los medicamentos los antibióticos se vuelven ineficaces y las infecciones se vuelven difíciles o imposibles de tratar.
Las secuencias genómicas de las bacterias son la clave para saber si estas podrán combatirse con ciertos antiobióticos o no. Dada su importancia para la salud pública, sus perfiles se han agrupado en la primera biblioteca española de bacterias resistentes a los antibióticos.
El Centro Nacional de Análisis Genómico (CNAG), Roche Diagnostics y el Complejo Hospitalario Universitario A Coruña-Instituto de Investigación Biomédica A Coruña (INIBIC) han sido los encargados de poner en marcha esta nueva base de datos digital gracias a una nueva metodología que permite obtener genomas bacterianos completos de manera más rápida y a gran escala.
El portal inCREDBle, que es el nombre de esta biblioteca de ADN disponible para consultas, exploración y descargas, recopila un total de 461 cepas bacterianas resistentes a los antibióticos, recogidas de 41 hospitales situados en 13 regiones diferentes de España, entre ellas Andalucía.
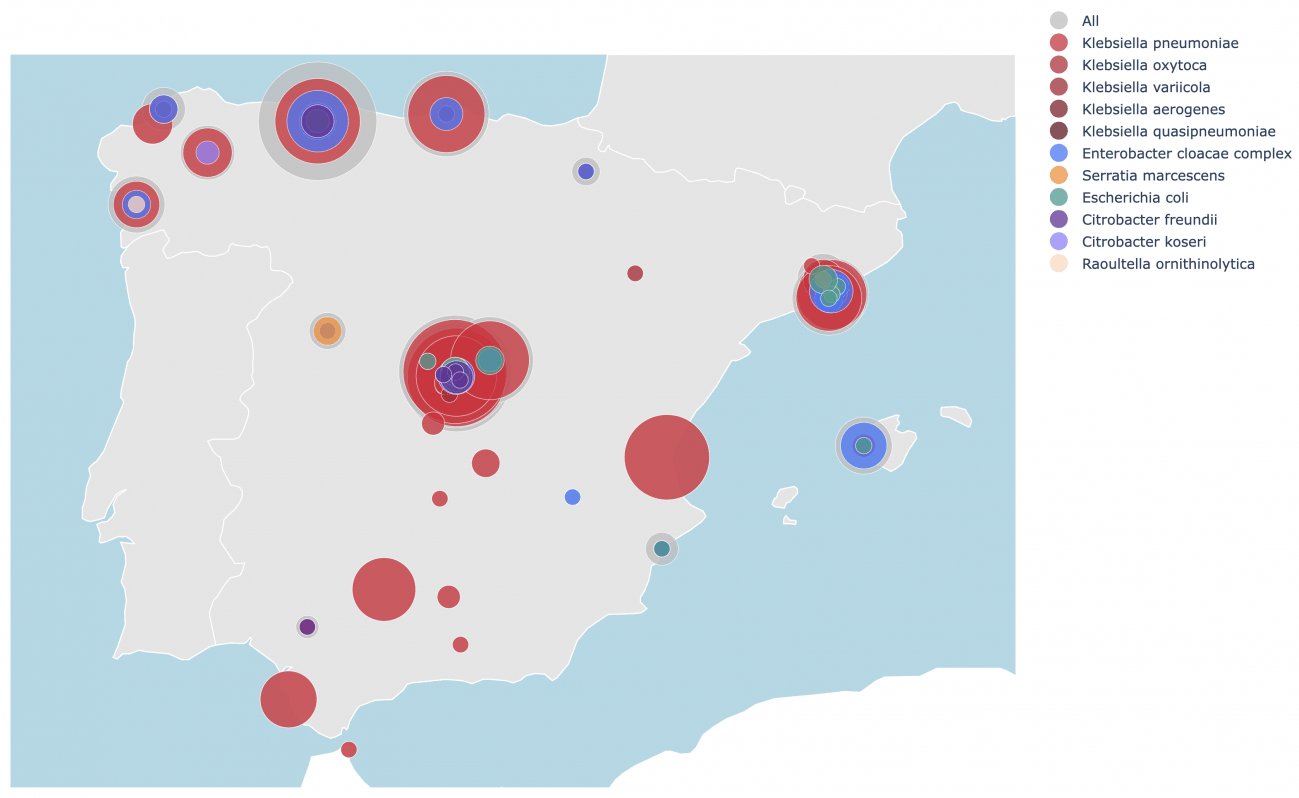
Distribución de las bacterias resistentes a los antibióticos por localización geográfica a nivel nacional. Imagen: CNAG.
La nueva base de datos digital complementa el perfil genómico de estas bacterias con datos clínicos, geográficos y microbiológicos, y con otros estudios online relacionados con la resistencia antimicrobiana, convirtiéndose en el recurso más completo para estudiar las enterobacterias, una de las familias más resistentes a los antibióticos.
Clave para futuros tratamientos
Según Tyler Alioto, autor del estudio y líder del equipo del CNAG, “la caracterización completa de las bacterias solo se puede lograr con ensamblajes completos del genoma que proporcionen el cromosoma principal, además del conjunto de plásmidos y su número de copias”.
“Para demostrar que esto se puede hacer a gran escala, secuenciamos 500 bacterias resistentes a los antibióticos con una combinación de tecnologías genómicas de última generación y desarrollamos un flujo de trabajo automatizado para procesar los datos”, añade.
El principal objetivo es proporcionar más información sobre los mecanismos por los cuales las especies del orden Enterobacterales adquieren o desarrollan esta resistencia a los medicamentos, así como también de los mecanismos subyacentes de diseminación.

Autores del estudio del Centro Nacional de Análisis Genómico. De izquierda a derecha (de arriba a abajo): Ivo Gut, Tyler Alioto, Marc Dabad, Eloi Casals, Fernando Cruz, Marta Gut, Jèssica Gómez-Garrido. Imagen: CNAG.
“Una de las estrategias que propone el Centro para el Control y Prevención de Enfermedades (CDC) en EE UU para la vigilancia de la resistencia antimicrobiana es el monitoreo de patógenos resistentes o multirresistentes. Justamente la base de datos genómica que hemos creado permite la identificación, localización y seguimiento de las bacterias gramnegativas que portan uno de los mecanismos de resistencia a antibióticos más peligrosos, las enzimas llamadas carbapenemasas”, afirma Germán Bou, autor del estudio.
“El trabajo puede servir para futuros estudios con un mayor número de cepas y que incluyan una mayor diversidad de patógenos resistentes. Esto permitiría una información dinámica y a tiempo real sobre la transmisión de microorganismos multirresistentes en nuestro país”, destaca Bou, responsable del INIBIC.
El estudio proporciona nuevos datos relacionados con la estructura de los plásmidos, precisamente por su papel importante en la transmisión de la resistencia antimicrobiana. Un conocimiento profundo de estos mecanismos podría ayudar a producir tratamientos más efectivos que eviten dicha resistencia.
“Las bases de datos como esta son realmente importantes para comprender la genética y biología con las que algunas de estas bacterias actúan contra los antibióticos. Son una herramienta muy valiosa para la investigación; ayudan en el desarrollo de nuevos métodos de diagnóstico y terapias de alto impacto para estos peligrosos microorganismos”, continúa Miguel Álvarez-Tejado, de Roche Diagnostics.
Referencia:
Alioto, Tyler S., et al. ‘Development of a Novel Streamlined Workflow (AACRE) and Database (inCREDBle) for Genomic Analysis of Carbapenem-Resistant Enterobacterales’. Microbial Genomics, 2023.
Suscríbete a nuestra newsletter
y recibe el mejor contenido de i+Descubre directo a tu email