Tomografía Electrónica para la visualización en 3D de la organización molecular de la célula

Las células están divididas en distintos compartimentos (p.ej. orgánulos) o regiones caracterizadas por determinadas propiedades (p.ej. concentración de determinadas proteínas). La morfología de estos compartimentos y regiones, su distribución espacial y la interacción entre ellos constituyen la arquitectura subcelular, que está íntimamente relacionada con el funcionamiento y especialización de la célula. Así, las anomalías en la arquitectura subcelular pueden reflejar o derivar en alteraciones funcionales. Por tanto, la identificación y caracterización de estas alteraciones estructurales puede proporcionar conocimiento sobre las bases patogénicas de enfermedades y contribuir a la búsqueda de nuevos desarrollos terapéuticos.
La microscopía electrónica es una técnica que clásicamente ha permitido avanzar en el conocimiento de la ultraestructura célular. En la última década, la tomografía electrónica (TE) se ha convertido en una técnica importante para la visualización y el análisis tridimensional (3D) de la arquitectura subcelular y de la organización molecular de células y tejidos in situ con un nivel de resolución de unos pocos nanómetros, permitiendo el abordaje de problemas fundamentales en biología celular y molecular.
Una fase clave dentro de la TE es la preparación de las muestras. La mejor preservación estructural -casi nativa- se obtiene mediante criofijación, en la que la muestra se congela de forma ultrarrápida (ms) y se mantiene hidratada en hielo amorfo (vítreo). Así se evitan los fijadores químicos clásicos y los artefactos que éstos introducen. La muestra se mantiene congelada así durante la observación al microscopio electrónico. De ahí el término crio-tomografía, que es una extensión de la crio-microscopía electrónica, la técnica que fue galardonada con el premio Nobel de Química 2017. En otros casos, la muestra se deshidrata para su visualización por TE a temperatura ambiente.

Crio-Microscopio Electrónico de última generación con capacidad para adquisición de datos tomográficos, disponible en el CSIC.
Los fundamentos de la TE son similares a los de la tomografía axial computerizada (TAC) comúnmente empleada en Medicina, pero haciendo uso de un microscopio electrónico y trabajando a escala nanométrica.
Esta técnica se basa en la adquisición de una serie de imágenes de la muestra tomadas a distintos ángulos de inclinación mediante un microscopio electrónico. Estas imágenes vienen a ser equivalentes a ‘radiografías’ de la muestra a distintas vistas. A partir de estas imágenes, se realizan un conjunto de procesos computacionales que culminan con la obtención de un volumen (o tomograma). Este volumen puede ser visualizado y analizado en 3D con el fin último de extraer información cuantitativa. Las técnicas de procesamiento de imagen y visión por computador juegan, por tanto, un papel fundamental en los estudios estructurales por TE (Figura 2). Muchos de estos procesos conllevan un tiempo de cálculo elevado, y la computación de altas prestaciones se ha convertido en una herramienta de gran importancia para proporcionar soluciones en tiempos razonables.
Los grupos de José-Jesús Fernández (Consejo Superior de Investigaciones Científicas, CSIC) y Ester Martín Garzón (Universidad de Almería, UAL) trabajan estrechamente desde hace bastante tiempo en nuevos desarrollos metodológicos para TE y su aplicación en la investigación de problemas de importancia biológica y biomédica. Conjuntamente desarrollan métodos avanzados de procesamiento de imagen 3D para el cálculo de volúmenes, filtrado de ruido, segmentación de componentes y análisis espacial de macromoléculas en su contexto celular. El aprovechamiento óptimo de la capacidad de cálculo de los computadores multinúcleo actuales y de las tarjetas gráficas es una de las áreas que lidera el grupo de la UAL a nivel nacional, y es clave para la ejecución eficiente de estos métodos.

Infraestructura de computación de altas prestaciones del grupo de la Universidad de Almería.
El grupo del CSIC, por su parte, aplica toda esta metodología para el estudio de la arquitectura subcelular neuronal y sus alteraciones en procesos neurodegenerativos. Otra línea de trabajo con esta metodología es la estructura del centriolo, un orgánulo implicado en la división celular y en la formación de cilios, cuyas mutaciones están detrás de diversas enfermedades humanas, como algunos tipos de cáncer.
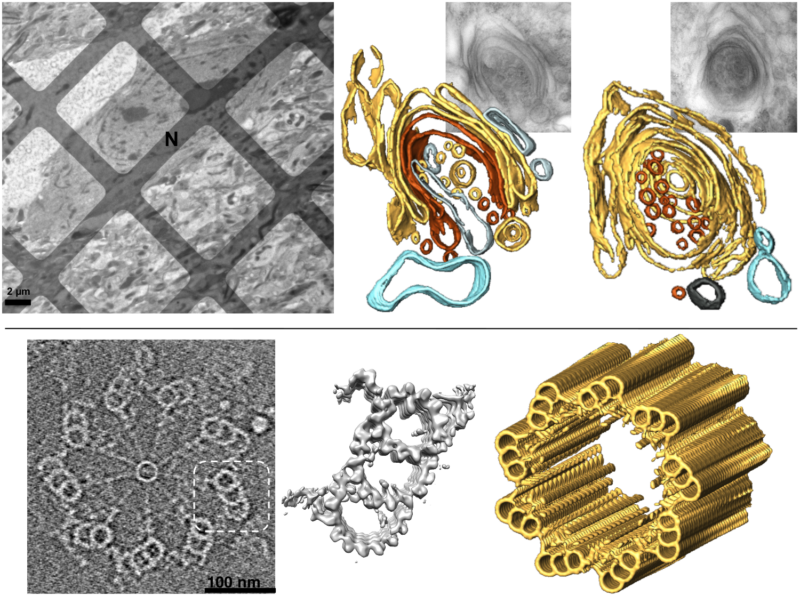
Arriba: Estudio de la arquitectura subcelular neuronal e identificación de nuevas estructuras in situ. Imagen de una neurona (N) de estriado de ratón (izquierda) y visualización 3D del Aparato de Golgi atrapando material citoplasmático superfluo o dañado para su posterior degradación.
Abajo: Centriolo. La TE ha permitido obtener información de la arquitectura molecular del centriolo y de su mecanismo de ensamblaje.
El uso combinado de tomografía electrónica, procesamiento de imagen y computación de altas prestaciones tiene un potencial enorme para el análisis de la arquitectura molecular in situ de sistemas biológicos complejos y heterogéneos. La sinergia de grupos multidisciplinares es importante para sacar el máximo provecho de todas facetas involucradas en estos estudios. Andalucía está bien situada a nivel nacional en el desarrollo de estas metodologías y su aplicación a problemas complejos tanto en ciencias de la vida como en ciencias de materiales.
Suscríbete a nuestra newsletter
y recibe el mejor contenido de i+Descubre directo a tu email




